华大在线讯(通讯员 张晓飞)近日,数学与统计学学院张晓飞教授领导的课题组在空间转录组学领域研究中取得了重要进展,在《Genome Biology》期刊发表了题为“ENGEP: advancing spatial transcriptomics with accurate unmeasured gene expression prediction”的研究论文(https://doi.org/10.1186/s13059-023-03139-w)。张晓飞为该论文的通讯作者,2022级博士研究生杨诗童为该论文的第一作者,华中师范大学为该项研究的独立完成单位。
在单细胞测序技术中,由于细胞在测序过程中的解离,导致无法获取细胞空间位置的相关信息,从而制约了我们对多细胞生物中细胞协调情况的深入理解。为了克服这一困境,空间转录组技术应运而生,为研究人员提供了一种能够同时捕捉细胞的基因表达和位置信息的新途径。然而,尽管基于成像的空间转录组方法具备高分辨率和高灵敏度,但它却面临着同时测量目标基因数量较少的挑战。
为了解决基于成像的空间转录组方法中目标基因数量有限的问题,张晓飞教授团队开发了一款名为ENGEP的计算方法。该方法利用k近邻加权回归和集成学习策略,能够准确预测空间转录组中未测基因的表达。ENGEP利用多个参考数据集、各种相似性度量和不同的近邻数量生成多个基础预测结果,并通过加权集成这些结果,形成了一致性的最终预测结果(图a)。这一策略不仅提升了预测的准确性和方法的稳健性,还避免了繁琐的参数选择过程,使用户更易于操作。
研究评估了ENGEP在三个使用不同技术生成的空间转录组数据集上的性能,并将其与五种前沿方法进行了比较。结果显示,ENGEP在准确预测基因表达方面显著优于其他方法,同时能够校正低质量基因的表达。此外,ENGEP还能够准确预测空间未测基因的表达模式,对增强空间转录组学数据具有重要意义(图b)。研究还揭示了在预测的未测基因中存在新的空间表达模式,为理解大脑、肿瘤等复杂组织的功能结构提供了新的洞见(见图c)。
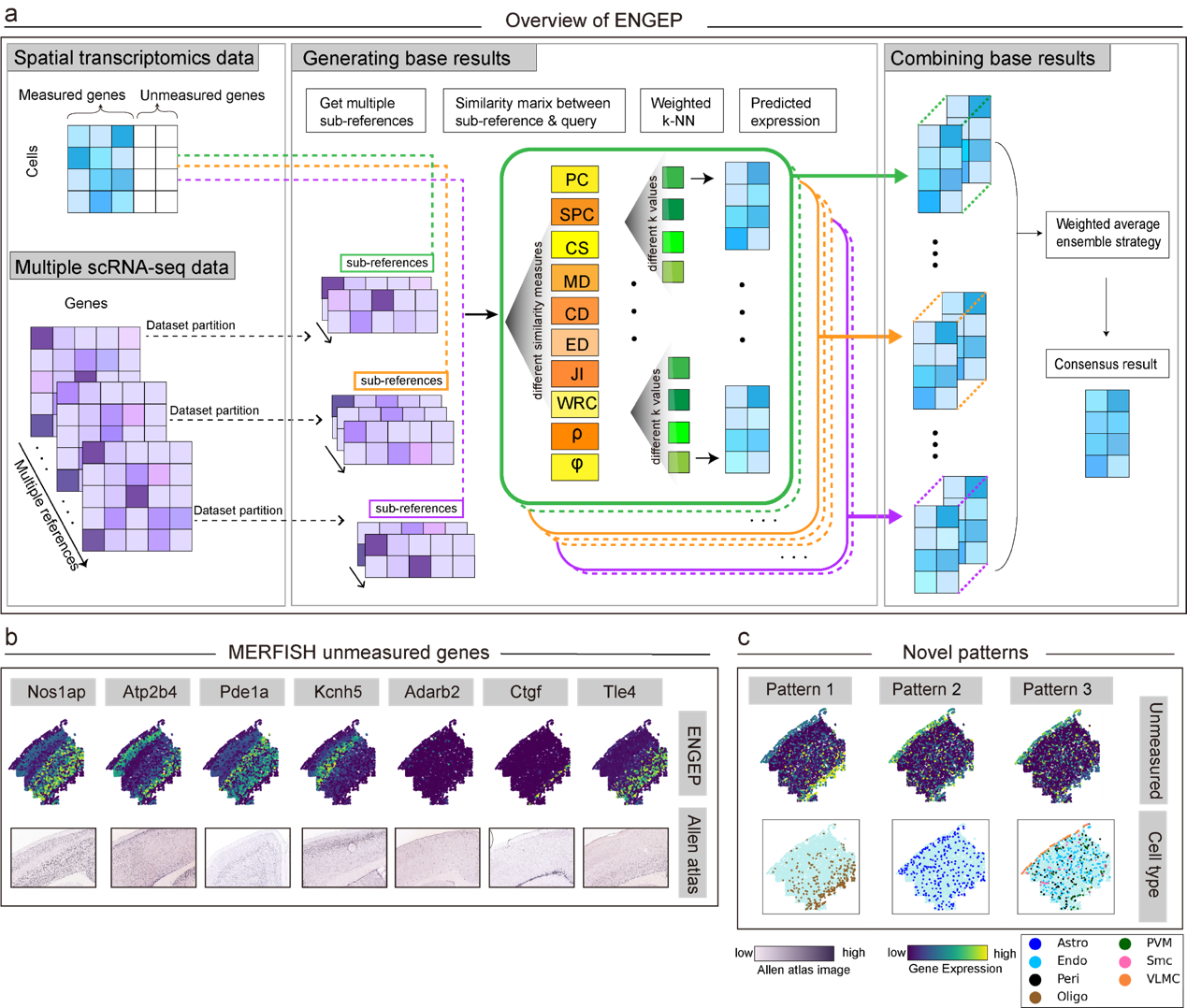
图为ENGEP增强空间转录组学数据。a:ENGEP算法工作流程。b: ENGEP准确预测空间未测基因的表达模式。c:ENGEP揭示新的空间表达模式。ISH图片来自Allen Mouse Brain Atlas (http://mouse.brain-map.org/)
总体而言,ENGEP成功解决了空间转录组学数据中未测基因较多的问题。相较于现有方法,ENGEP的独特之处在于不依赖于单一的参考数据集或方法,而是整合了多样性的结果,克服了以往方法的局限性,实现了更一致和准确的预测。其集成学习策略为参考数据集的选择、批次差异的校正以及相似性度量的选择提供了新的思路。此外,相对于其他方法,ENGEP在运行时间和内存使用效率上也表现出色,使其能够轻松应对大规模数据集的分析。该工作得到了国家自然科学基金的资助。
(审读人:王海 郭玉劲)