华大在线讯(通讯员 朱潇潇)近日,物理科学与技术学院赵蕴杰教授与美国达特茅斯计算机学院Soroush Vosoughi教授、美国乔治华盛顿大学曾辰教授合作在软凝聚态与生物物理领域取得了新的研究进展,在《Nature Communications》(影响因子:17.694)发表题为“Evaluating native-like structures of RNA-protein complexes through the deep learning method”的研究论文。我校直博生曾成伟为该论文的第一作者,达特茅斯计算机学院简弋人为共同第一作者,物理科学与技术学院赵蕴杰教授为论文的通讯作者,华中师范大学为第一完成单位。
RNA-蛋白质复合物是生命体中广泛参与生命活动的软物质分子,精确的三维结构有助于理解其调控机理与相关疾病的治疗。X射线晶体衍射、核磁共振或冷冻电镜等实验方法解析RNA-蛋白质复合物结构需要花费大量的时间和财力,亟需发展RNA-蛋白质复合物结构预测的理论计算方法。基于统计势能的评分函数是目前常用的RNA-蛋白质复合物结构预测评估模型,在刚性对接问题中表现较好,柔性对接的结构评估仍然没有解决,依然是极具挑战性的难题。RNA-蛋白质复合物界面相互作用包括静电、范德华、碱基堆积与氢键等多种类型并形成相互作用网络,RNA分子在复合物结合前后有一定的构象变化,需要发展适用于描述分子复杂系统的多尺度、多维度复杂动力学行为的统计物理新模型。
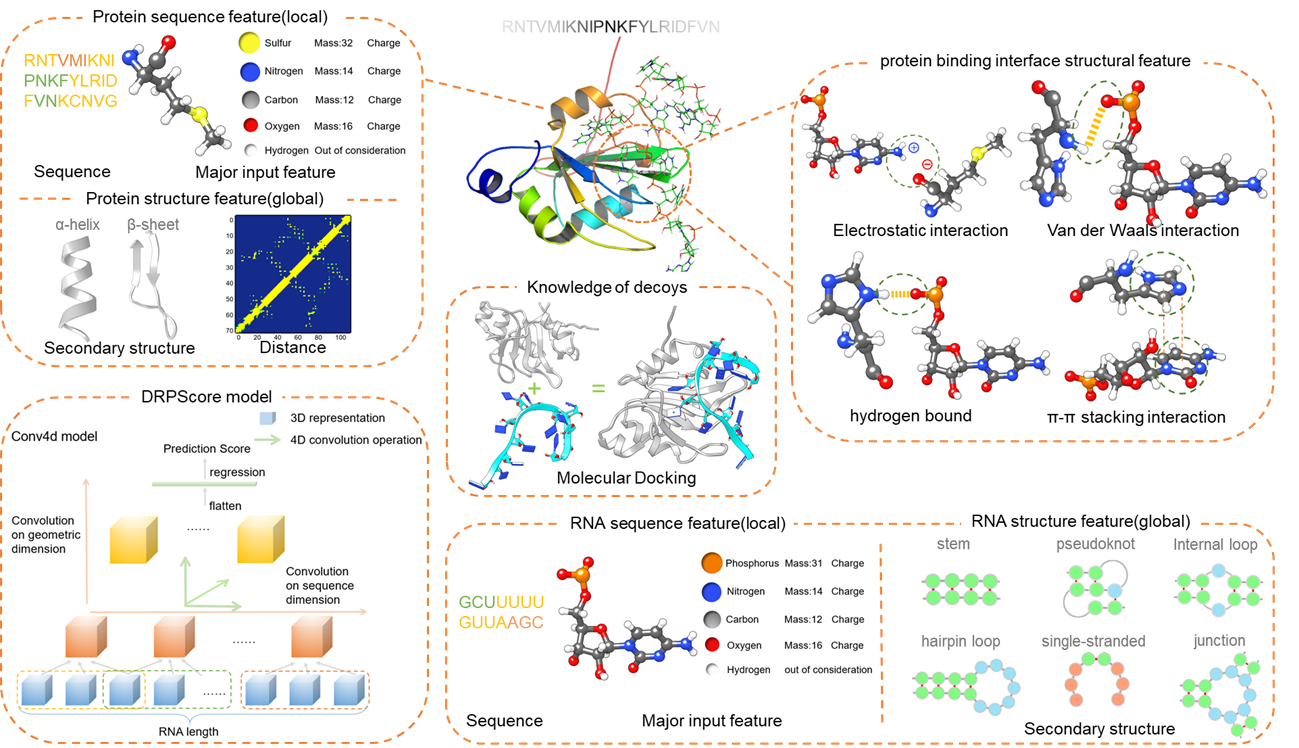
图1. DRPScore模型训练中学习的RNA-蛋白质复合物特征与4DCNN深度学习模型
赵蕴杰教授课题组发展了基于人工智能和统计物理模型相结合的新方法DRPScore(图1)。该方法通过分子对接模拟复合物结合的构象变化,可以考虑柔性对接中分子结构的变化。另一方面,该方法聚焦于RNA-蛋白质相互作用界面,通过4DCNN模型不仅可以有效学习原子质量、原子电荷、相互作用类型(静电相互作用、氢键等)和相互作用距离的局部特征,还可以学习复合物二级结构相互作用(RNA为配对、发卡环和内环等;蛋白质为螺旋和beta折叠单元)与口袋拓扑结构等全局特征。
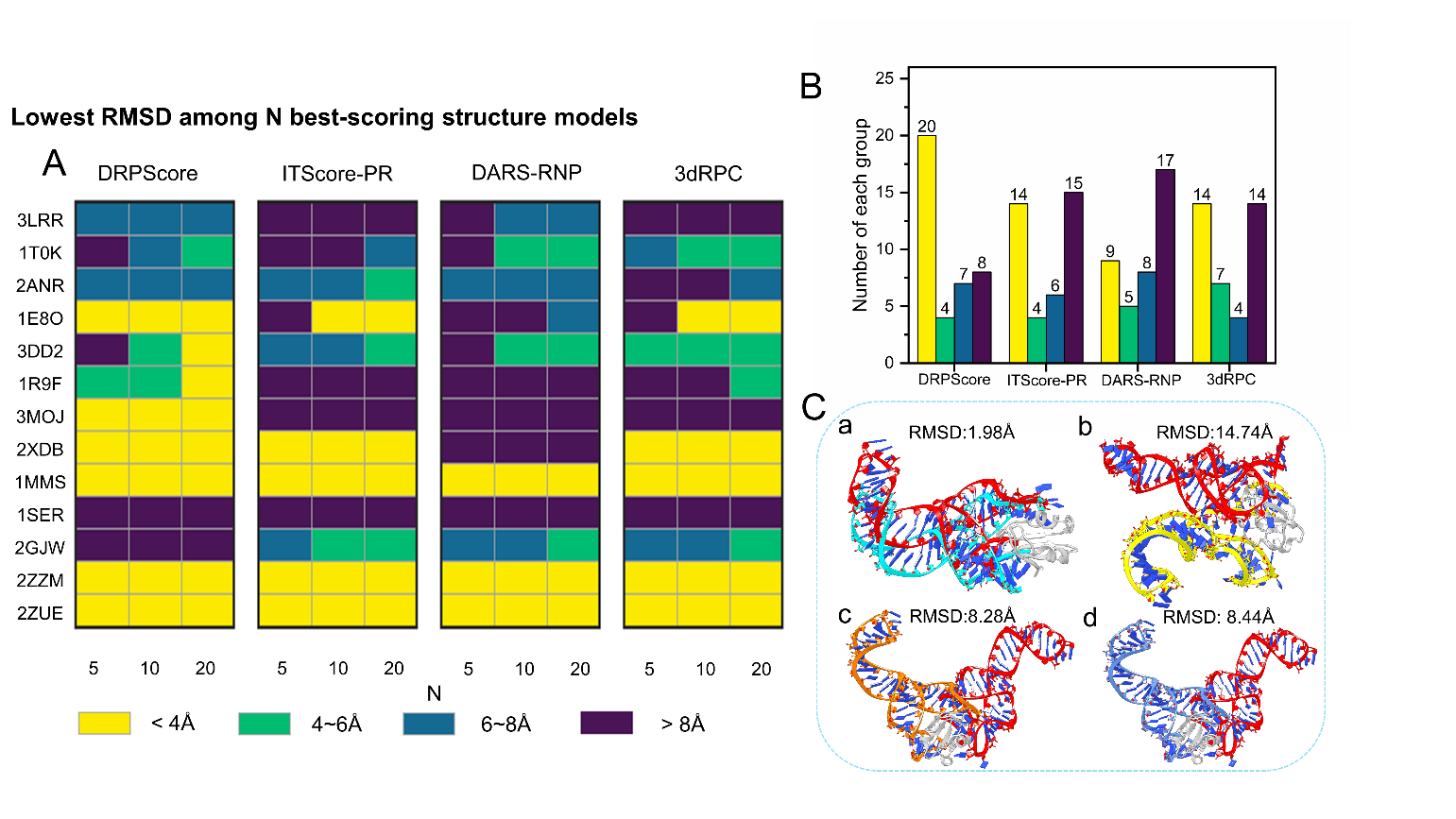
图2. 柔性对接测试集结果。黄色为近天然态结构,DRPScore的预测精度显著高于传统模型
柔性对接是RNA-蛋白质复合物结构预测中尚未解决的瓶颈问题。例如,复合物界面相互作用面较小时,传统统计势模型过于敏感并导致较难选择近天然态结构。DRPScore可以通过较少的相互作用数据学习RNA-蛋白质复合物界面特征,较传统方法有显著的提升(图2)。DRPScore在不同柔性程度的测试集上进行了广泛评估,RNA-蛋白复合物结构预测的刚性对接测试集中成功率为91.67%,柔性对接测试集中的成功率为56.14%,较传统方法提高了10.53-15.79%,有效解决了结构评估中由柔性诱导的构象变化问题,显著提高了RNA-蛋白质复合物结合界面相互作用预测的准确性。DRPScore是软凝聚态与生物物理研究领域中研究生物大分子的有效工具,将对RNA相关复合物结构预测、结构设计与相关药物开发有重要作用。
该研究工作得到国家自然科学基金面上项目、湖北省百人计划和华中师范大学优秀青年团队等资助。
全文链接:https://doi.org/10.1038/s41467-023-36720-9
(审读人:俞云伟)